

中国科学院系统生物学重点实验室吴家睿教授和曾嵘教授目前在国科大杭州高等研究院筹建科学家工作室,工作室联合中国科学院分子细胞科学卓越创新中心,海军军医大学第一附属医院(上海长海医院)等单位,9月3日在国际学术期刊Cancer Cell在线发表题为“Integrated Omics of Metastatic Colorectal Cancer”的研究论文。该研究工作整合系统生物学理论,组学分析技术和临床基础,完成了首个中国人结直肠癌转移相关的多组学整合图谱,提出了转移性结直肠癌分子分型与个性化治疗新策略。

近年来随着手术、靶向和免疫治疗的快速发展,早期癌症患者的生存率得到很大改善,但晚期转移肿瘤的治疗仍是普遍难题。最近的泛癌(Pan-Cancer)基因组研究表明,原位肿瘤基因组和转移肿瘤基因组之间的差异甚小。该结果进而引出了一个急需回答的问题:肿瘤转移相关的特征体现在哪些分子层面?此外,目前大多数靶向药物治疗是基于基因突变进行筛选的,没有相关基因组特征的患者可能因此失去治疗机会,这对晚期转移肿瘤患者无疑意味着更差的预后。显然,要解决这些难题与挑战,需要开展基于系统生物学的肿瘤转移精准医学研究。
为此,研究团队系统性地收集了转移性结直肠癌患者480个临床样本,包括原发灶与相应的转移灶,近端癌旁组织及远端正常组织;然后对这些样本分别进行了全外显子测序、甲基化芯片、定量蛋白质组和磷酸化蛋白质组等多组学数据采集。通过这些临床大数据分析以及与西方人数据集的比较,研究人员绘制了首个中国人结直肠癌及转移相关的多组学整合图谱。
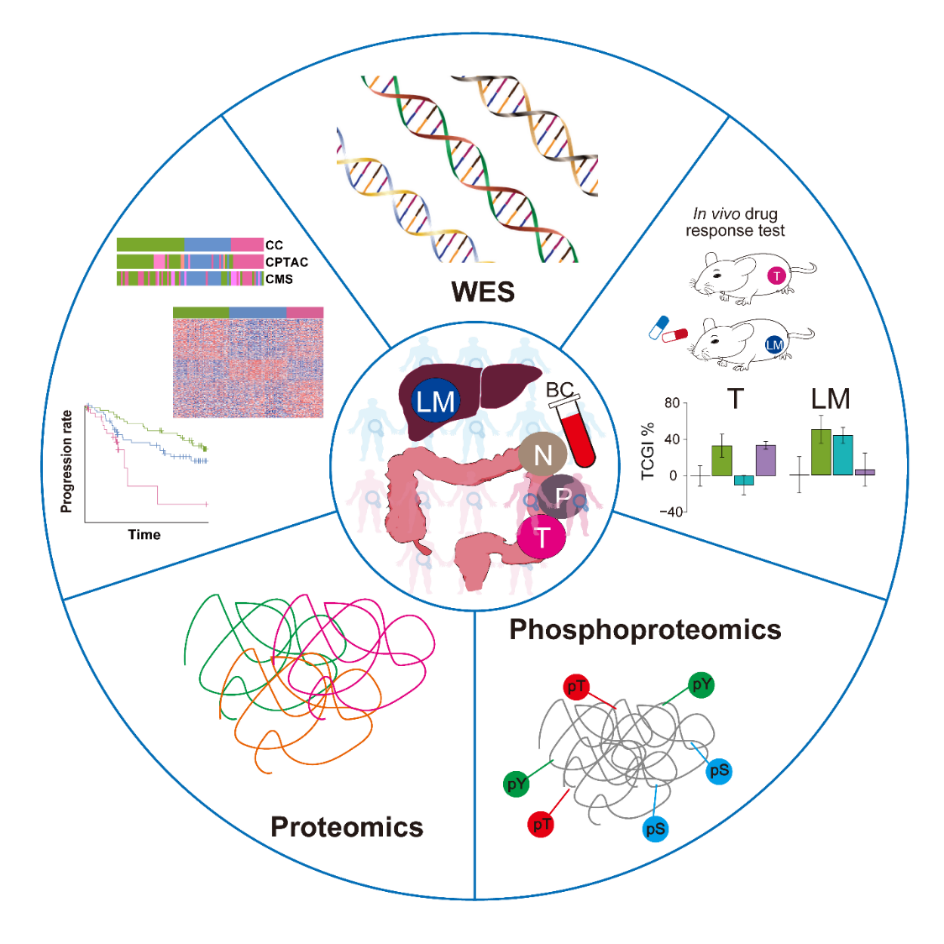
绘制首个转移性结直肠癌多组学整合图谱
此项研究最突出的成果是,首次系统地揭示了中国人结直肠癌与转移的分子特征谱。首先,通过定量蛋白质组数据分析,划分出了三个具有显著不同功能偏好的结直肠癌分子亚型,三类亚型的预后有显著差异;特别的是,基于定量蛋白质组的三分型,仅在晚期癌症患者中,也能有效区分预后,这为晚期癌症患者临床决策和及时治疗提供了重要指标。其次,基于原发灶的磷酸化蛋白质组定量谱,能够有效判别结直肠癌患者的转移情况,提示蛋白质磷酸化调节的异常与肿瘤转移高度相关;研究还发现同一患者转移灶相比于原发灶在基因组水平上并没有明显差异,这与近期Nature发表的一项涵盖2520例全基因组测序数据的转移性泛癌研究结果一致,提示肿瘤转移灶和原发灶的基因组特征非常相似。更重要的是,转移性结直肠癌的转移灶与原发灶在蛋白质、磷酸化修饰、激酶底物网络水平上却明显不同,表明对于转移性肿瘤的分析需要同时考虑原发灶和转移灶状态。该多组学数据整合研究为加深对中国人群和转移性结直肠癌的理解提供了丰富的资源。

首个中国人转移性结直肠癌(Chinese colorectal cancer,CCRC)蛋白质分子分型
此项研究的另外一个重要成果是突破精准医学靶向治疗对基因突变的依赖,直接针对作为药靶的蛋白质进行筛选。研究人员首先基于配对的原发灶-转移灶的蛋白质组/磷酸化蛋白质组数据,量化分析了激酶与底物相互作用网络;通过激酶活性富集、激酶底物相关性网络分析了蛋白质亚型之间,原发灶和转移灶之间的异质性。在此研究基础上,研究人员建立了来自22位结直肠癌患者的31个人源性肿瘤异种移植模型,并进行了三种激酶靶向药物(Afatinib、Gefitinib和Regorafenib)的药敏实验,验证了来自同一患者的原发灶和转移灶对同一药物表现出不同的反应。研究结果表明,对于转移性肿瘤的治疗不仅是个性化的,甚至是位置特异的。更重要的是,在药敏模型对应的肿瘤组织中,三种药物靶向基因几乎没有发生相关突变,然而,一些肿瘤组织却显示出良好的药物敏感性,这表明在指示药物敏感性方面,激酶底物相关性网络的判别可能比基因突变存在与否更为敏感和直接。通过构建激酶-底物网络的机器学习模型,该研究为药效判别提供了准确的预测,从而为癌症个性化用药提出了超越基因组分析的新方向。

基于in-vivo药敏测试和机器学习的靶向药物反应判别模型
在此工作基础上,研究团队正在构建更大规模的基于组学大数据肿瘤用药数据库,争取早日发布并在临床应用,最终使癌症患者获益。 此项研究工作在曾嵘研究员、张卫教授和吴家睿研究员共同指导下,主要由曾嵘组副研究员李辰、曾嵘组博士研究生孙怡迪、长海医院肛肠外科主治医师于冠宇、吴家睿组研究助理崔静如和长海医院肛肠外科副主任楼征等合作完成。该项目受到中国科学院战略性先导项目、科技部重点研发计划和自然科学基金委经费支持。